ANTIGENOS RECOMBINANTES DE SALSOLA
V.
del Pozo, E Civantos, J. Carnes*, I.
Cortegano, B. Cárdaba, S. Gallardo, A. Lezaun+, P. Palomino, E.
Fernandez-Caldas* , C. Colas+, C. Lahoz.
*C.B.F. Leti. Madrid
+ Servicio de Alergia.
Hospital Clínico Lozano Blesa. Zaragoza
RESUMEN
Salsola kali es una maleza de la familia
de las Quenopodiaceas, que se encuentra ampliamente distribuida en la costa
europea, norte de Africa, Australia y USA.
El
objetivo de este trabajo es la clonación del alergeno principal de Salsola kali, y su posterior expresión
en levaduras lo que permitirá obtener gran cantidad de proteína homogénea. Para
ello, la
proteína mayoritariamente reconocida por los sueros de pacientes alérgicos
(>50%), fue fragmentada con tripsina para obtener las secuencias de peptidos
internos, mediante espectrofotometría de masas MALDI-TOF.
A partir de la secuencia aminoacídica, se
diseñaron cebadores específicos que nos permitirán amplificar el ADN
correspondiente al antígeno mayoritario, a partir del ARN total del polen.
A partir de las secuencias internas
se han obtenido dos fragmentos de cDNA para Sal k 1: 500 pb y 650 pb. El
análisis comparativo de secuencia no ha demostrado homologías importantes con
ninguna proteína clonada conocida. Actualmente se esta uniendo estos dos
fragmentos, para así expresar la proteína recombinante en levaduras.
INTRODUCCIÓN
Salsola
kali o pestifer (1) pertenece taxonómicamente a la
familia de las Chenopodiáceas que junto con la familia de las Amarantáceas,
pertenecen al orden Centrospermales, superorden Monochaamydeae, subclase
Archichlamydeae, clase Dicotiledóneas. Dentro de la familia de las
Chenopodiáceas existen cerca de 110 géneros de los cuales los clínicamente más
importantes son Chenopodium y Salsola.
Es una maleza, denominada
comúnmente Salsola, pincho, espinardo, etc, de amplia distribución en regiones
templadas, siendo su hábitat suelos removidos, barbechos y áreas litorales. Están bien adaptadas a los suelos
ricos en sal, ya del litoral o del interior. Han sido descrito como causa
importante de polinosis especialmente en ambientes de baja pluviosidad de la
mitad oeste de EEUU, Irán, Arabia Saudí, Sur de Europa y norte de África
Son plantas fundamentalmente
anemófilas con un amplio periodo de polinización que va desde abril hasta
octubre con variaciones regionales dependiendo de las condiciones climáticas
Las chenopodiáceas producen, en general, una gran cantidad
de pólenes, lo que, junto con su ubicuidad y gran abundancia en ciertas áreas,
contribuye a su importante representación en los recuentos de pólenes de la
atmósfera en ciertas regiones. En algunas ciudades como Elche este polen puede
representar hasta el 18% de los pólenes recogidos, aunque la media se sitúa en
torno al 4-5% (2)
Destaca su gran resistencia a la
sequía y de hecho, en España hay 2 zonas donde la planta es especialmente
relevante: Valle del Ebro y área de confluencia de Almería, Albacete y Murcia,
ambas zonas semidesérticas, donde la pluviosidad es escasa.
A pesar de la relativamente baja
cantidad de polen recogido en los estudios aerobiológicos (aproximadamente un
5% de los pólenes totales recogidos, con picos de 50 granos/m3) son
la primera causa de sensibilización a polen en Toledo y Elche, la segunda en
Logroño, la tercera en Zaragoza y Ciudad Real y la cuarta en Málaga y Sevilla
(3), por lo que se considera que se trata de pólenes “agresivos”, cuyo umbral
de reactividad ha sido estimado en 10-15 granos/m3 (para generar
sensibilización en el 46% de polínicos) (2).
Sin embargo, a pesar de su
reconocida alergenicidad, se conoce muy poco sobre los alergenos de este polen.
En 1981 (4) se aislaron dos glicoproteínas, RT1 y RT2, con PM de 39 y 42 kDa y
pI de 6.7 y 6.2 respectivamente, que en la actualidad se consideran el mismo
alergeno y se denomina Sal p 1. Por técnicas de “inmunoblotting”, Lombardero et
al (5) observaron 2 bandas proteicas que fijaban IgE, una de 14 y otra de
35kDa. Posteriormente, de la Hoz (7) tras la separación de los componentes
proteicos de S. kali identificó 28
bandas diferentes con PM de 13-106 kDa, pero, a pesar de esta heterogeneidad,
un 64% fijaron IgE en la zona de 54 kDa y un 44% en una de 36.
Gracias a los avances en el campo de
las técnicas de Biología molecular, se puede hoy día obtener alergenos
recombinantes en gran cantidad y con características muy similares a las
nativas, que permiten disponer de nuevas herramientas útiles para el
diagnóstico.
MATERIALES Y METODOS
Aislamiento de RNA
La extracción de RNA se realizó a partir del polen de Salsola kali. Se lisa el polen con solución de lisis (8M hidrocloruro de guanidinio, 20mM MES y 20mM EDTA). La mezcla se congeló y descongeló con nitrógeno líquido y el homogeneizado se pasó por las columnas QIAshredder. El RNA total se extrajo con el Rneasy kit (QUIAGEN).
Secuenciación de Sal k 1
Las proteínas del extracto del polen de Salsola kali, se separaron en un SDS-PAGE y a continuación se transfirieron a una membrana PVDF. Se obtuvo una proteína de 41 kDa que se corresponde con el antígeno mayor de S. kali y se cortó de la membrana para su posterior secuenciación. La secuenciación de peptidos internos se realizó por espectofotometría de masas MALDI-TOF (8).
Síntesis de cDNA por 3´RACE y
5 ´RACE
La síntesis de cDNA se realizó mediante el sistema 3´RACE ( Rapid Amplification of cDNA ends method, GIBCO BRL, USA) según el protocolo.
A partir del cDNA total se hizo una PCR con un primer diseñado a partir de un péptido obtenido de la secuenciación interna de la proteína, Salka 1 y el UAP (primer universal de amplificación). La PCR se realizó en 35 ciclos con una temperatura de alineamiento de 50ºC.
Para la obtención del fragmento de Sal k 1 que faltaba se utilizó el sistema 5´RACE. Este consiste en a partir de mRNA del polen de Salsola kali, con un primer kira 1.1 sintetizar el cDNA hacia el extremo 5´. Para obtener un fragmento de cDNA del que se conozcan sus extremos hay que añadirle una cola poli C y con un primer que la reconoce y el que nosotros hemos diseñado (Salka 1) amplificar por PCR esta región. La PCR se realiza en 35 ciclos con una temperatura de alineamiento de 50ºC.
Clonaje y secuenciación del
cDNA de Sal k 1
Los fragmentos de PCR se purificaron de un gel de agarosa low melting al 1% utilizando el sistema PCR Preps DNA Purification System (PROMEGA). Estos fragmentos de PCR se secuenciaron y se compararon en el GenBank para ver si presentaban homología con alguna secuencia ya existente.
RESULTADOS
Para caracterizar las proteínas integrantes del polen de Salsola kali se sometieron a un
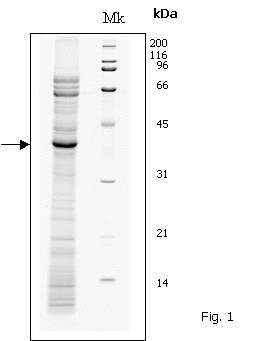
SDS-PAGE, en el cual se separaron las proteínas. En la figura 1 se observan
todas las proteínas del extracto del polen de Salsola kali. La banda que aparece más intensa es el antígeno
mayoritario que está a una altura de 41 kDa.
![]()
![]()
Se realizó la transferencia de las proteínas del polen de Salsola kali a una membrana PVDF, de la que se puede, cortar la banda que corresponde con el peso molecular de la proteína que queremos secuenciar. Esta proteína, el antígeno mayoritario, tiene el extremo N-terminal bloqueado con lo que no se ha podido secuenciar por completo. Mediante espectofotometría de masas (MALDI- TOF) se obtuvieron pequeños péptidos, cuya localización dentro de la proteína nos es desconocida, con los que hemos diseñado cebadores específicos.
Los péptidos que se obtuvieron se muestran a continuación con los cebadores que se diseñaron:
1.- Pro
Tre Iso Tre Iso Gly Gly Pro Glu Tyr Arg
Val Leu
Leu
2-. Tre
Iso Phe Phe Asp Ala Tyr Lys
Leu Met
3.- Gly
Treo Ser Tyr Ile Val Ile Lys
4.-Glu pro Ala Glu Glu Phe Treo Treo Ile Ser Asp Ala Val Lys
Basándonos en estas secuencias, se diseñaron unos cebadores específicos que nos permitieran clonar en romo y en cohesivo introduciendo una secuencia de corte para Sal I y Spe I.
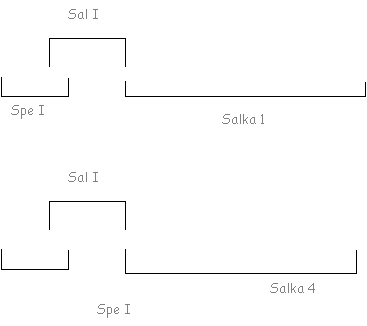
GGG ACT AGT CGA CGG NGG NCC NGA RTA YMG N
GGG ACT AGT CGA CGA RCC NGC NGA RGA RTT Y
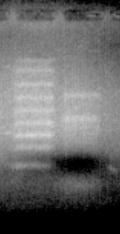 Para la obtención del cADN en primer lugar se aislo el
ARN total del polen de Salsola kali, la figura 2 A muestra una microfotografia del
polen de Salsola Kali. La figura 2B muestra el ARN total obtenido tras romper
los granos de polen. Tres bandas diferentes se pueden observar que corresponden
al ARN 28S y 18S. la banda 5S no se ve bien debido a su pequeño tamaño.
Para la obtención del cADN en primer lugar se aislo el
ARN total del polen de Salsola kali, la figura 2 A muestra una microfotografia del
polen de Salsola Kali. La figura 2B muestra el ARN total obtenido tras romper
los granos de polen. Tres bandas diferentes se pueden observar que corresponden
al ARN 28S y 18S. la banda 5S no se ve bien debido a su pequeño tamaño.
5
S
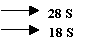
![]()
FIGURA 2 A FIGURA 2 B
A partir de este ARN total se hizo el cADN con el primer
Salka 1 y Salka 4 probaron y se obtenía una secuencia de un tamaño de 650 pares
de base, un tamaño considerable para ser parte de Sal k 1.
E
l fragmento que se obtuvo se muestra en la figura 3.
Figura
3

![]()
La banda es sólo una parte del cDNA que codifica para el
antígeno mayoritario de Salsola kali.
La banda de ADN se extrajo del gel de agarosa de bajo punto de fusión ( Bio-Rad Richmond, Ca, USA) utilizando el Wizard PCR preps DNA purification System (promega Madison, WI, USA) posteriormente se fosforiló con la enzima T4 polinucletido Kinasa ( Promega) y se insertó en el sitio Sal 1 del plásmido Bluescript (Stratagene, La Jolla). Las bacterias E. coli XL-1 blue MR supercompetentes se transformaron con el ADN, y se seleccionaron los clones positivos para secuenciar.
La secuencia de pares de bases obtenida se ha
introducido en el Genbank con nº de acceso: AF449490. A continuación se muestra
la secuencia:
5´TATTCATTCGGAATCATCTTGCTTTCAGTTGCTCACAGCAAGGCCTCCTATGGCATTGTCCTACCATGTGGA
AGAAGCCATTGATGCGGGTAACTTTAAAGAGGGTACTAGATCCTTCTATCTCCGATTGGCCTGTCCAAGAGG
CTTTGTCATTAGCTCAATTGGATATCAAGTGTTGCGAACTTAGGAAGAAGGATAGGCCCAACTTNGACTCGG
AAATCATGCCAGAGCTGGTCCGGTTACGAGACTTAGCCTTGAGTTCTTGATCTTTCTCTTTGCTTTTCTTTTCT
TTTTGGGG 3´
Haciendo
una comparación con otras secuencias en el Genbank, no se ha obtenido una
homología importante con nada que esté secuenciado hasta ahora, si bien es
cierto que ningún alergeno de la familia de las quenopodiaceas esta clonado.
Unicamente se ha encontrado un 28,53 % de homología con Entandrophrafma
cylindricum y <10% con Arabidopsis thaliana.
Para poder obtener el DNA que codifica para la proteína completa, se utilizó el sistema 5´RACE, con el fin de sintetizar la parte del DNA desde el extremo 5´hasta donde se une el primer Salka1. De este modo se tendría la cadena completa de cDNA y sólo habría que unirla. Por estimación el cDNA de Sal k 1 tendría más o menos 1200 pares de bases. El fragmento de DNA sintetizado por el 3´RACE tiene 650 pares de bases con lo que por este sistema deberíamos obtener un fragmento de aproximadamente 550 pares de base. Al hacer el cADN con el protocolo del 5´RACE, aparecen dos bandas a una altura que podrían corresponder con el fragmento que vamos buscando.
![]()
Actualmente estamos clonando el fragmento completo de ADN que codifica para Sal k1 en dos vectores: uno plásmidico que nos permita hacer la expresión de la proteína en reticulocitos de conejo, y en un vector que permita la expresión de Sal k 1 en Pichia pastoris. Esto nos permitirá comparar la actividad de estas proteínas con la proteína nativa.
BIBLIOGRAFÍA
1.
Weber,
R., Nelson, H.S. Pollen allergens and
their interrelationships. Clin Rev Allergy 1985; 3:291-318.
2.
Pola, J., Zapata, C., Sanz, E. Polen
de Chenopodiáceas. Comunicación de XXX Reunión de la Sociedad Andaluza de
Alergología e Inmunología Clínica. 20001:21-29.
3.
Subiza, J., Feo Brito F, Pola J et al. Pólenes alergénicos y polinosis en 12 ciudades españolas. Rev. Esp. Alergol Inmunol
Clin.1998;2:45-48.
4.
Shafiee A, Yunginger JW, Gleich GJ. Isolation and characterization of Russian
thistle (Salsola pestifer) pollen allergens. J Allergy Clin Immunol
1981;67:472-81.
5.
Lombardero M, Duffort O, Selles JG, Hernández
J, Carreira J. Cross-reactivity among
Chenopodiaceae and Amaranthaceae.
Ann Allery 1985:54:430-436.
6.
Gómez J, Mansfield LE, Frederick RW, Rael ED. Analysis of the individual allergens of
Russian thistle pollen by an
Enzyme-linked immunobltting technique. Journal Asthma 1989;
26:243-50.
7.
De La Hoz B. Rinoconjuntivitis y
asma por hipersensibilidad al polen de Salsola
Kali (Chenopodiaceae): Aspectos
clínico-inmunológicos y valoración de la inmunoterapia. Tesis Doctoral
Madrid.1995.
8.
Shevchenko A, Wilm M, Vorm O, Mann M. Mass spectrometric sequencing of proteins
silver-satiened polyacrilamide gels. Anal Chem 1996; 68 (5):850-858.