|
Inmaculada Cerecedo*, MC Dieguez*, Javier Zamora+, Belén de la Hoz*
*Servicio de Alergología. +Unidad de Bioestadística.
Hospital Ramón y Cajal. Madrid.
|
El objetivo fundamental
en el diagnóstico de las reacciones alérgicas a alimentos mediadas por
IgE es el establecimiento de una asociación causal entre el alimento y
las manifestaciones clínicas referidas por el paciente (que se recogen
mediante la historia y la exploración clínica inicial) y la
identificación del mecanismo IgE subyacente (mediante pruebas cutáneas
y/o determinación de IgE sérica específica). En muchos casos la
realización de estas pruebas precisa, para alcanzar el diagnóstico la
realización de una provocación oral (PO).
Las técnicas de diagnóstico disponibles en la práctica clínica habitual
en alergia se desarrollaron a lo largo del siglo pasado. A finales del
siglo XIX se proponen las pruebas cutáneas como método de diagnóstico
para las enfermedades alérgicas. Es a partir de los años 60, con la
caracterización de la IgE como inmunoglobulina desencadenante de la
respuesta alérgica, cuando se dispone de las primeras técnicas “in vitro”.
En los años 90 se clonan los primeros alérgenos, lo que nos lleva a
disponer de extensas librerías de alérgenos recombinantes para
diagnóstico. Este avance crea la necesidad de nuevas técnicas de
análisis masivo y simultáneo. El objetivo de esta nueva tecnología es
pasar del diagnóstico basado en extractos alergénicos (formados por
conglomerados de moléculas alergénicas y no alergénicas) al “diagnóstico
por componentes”, es decir, establecer exactamente la molécula
alergénica que es la causante de la reacción(1). La deficiente
estandarización de los extractos actuales (expresión de alergenos en
ciertas condiciones de maduración del alergeno, contaminación, infra-representación
o degradación de alérgenos importantes) y el identificar la fuente
alergénica, pero no las moléculas responsables de la reacción nos
dificulta la labor de diagnóstico, manejo y tratamiento de los
pacientes.
Un microarray o micromatriz es un soporte sólido plano (habitualmente un
cristal tratado) en el que se encuentran inmovilizadas de manera
ordenada (“arrayed”) cientos /miles de dianas. Este tipo de ensayos
cumplen cuatro características: son sustratos planos, en los que dianas
microscópicas (spots) son inmovilizadas formando filas y columnas y
existe además una unión específica entre estas dianas inmovilizadas en
el sustrato plano y moléculas en solución.
Tras la conclusión de la secuencia completa del genoma humano, de la que
se dispone de un borrador en 2001 y se completa a finales de 2004(2), es
necesario caracterizar funcionalmente 20.000-25.000 genes que codifican
proteínas. La información generada no es abordable con las técnicas
analíticas disponibles en ese momento, por lo el microarray surge como
el ensayo más adecuado para este trabajo. En el campo de la genómica se
han empleado los microarray en el conocimiento de las bases moleculares
de la fisiopatología de las enfermedades, la identificación de nuevos
genes marcadores (de diagnóstico, pronóstico, monitorización de la
evolución o de la respuesta terapéutica) y el desarrollo de fármacos. El
éxito del empleo de la tecnología microarray en el campo de la genómica
da lugar a su expansión y adaptación a otros campos como el estudio de
proteínas, reacciones enzimáticas o inmunológicas.
En el estudio de las respuestas alérgicas IgE mediadas, estos ensayos
tienen una aplicación diagnóstica directa, describiendo patrones de
reconocimiento inmunológico (distinción entre distintos grupos de
pacientes) e indirecta, mejorando el rendimiento de las pruebas
diagnósticas ya disponibles en la actualidad (conociendo epítopos se
podrían fabricar extractos que mejorasen el rendimiento de estas
pruebas). Una aplicación terapéutica potencial sería en el desarrollo de
IT específicamente dirigida a esas regiones inmuno-dominantes.
La alergia a proteínas de leche de vaca (APLV) afecta aproximadamente al
2.5% de los niños menores de 2 años. El tratamiento consiste en
instaurar una dieta de evitación y programar visitas regulares de
seguimiento en las que se evalúa la evolución. Aunque la mayoría de los
niños alcanza la tolerancia alrededor de los 4-5 años, el 15-20% de los
níños con APLV no ha superado la enfermedad en la segunda década de
vida(3-7).
La leche de vaca contiene 3g de proteína/100 mL e incluye al menos 25
proteínas distintas entre séricas y caseínas. Las proteínas de la leche
son de los alérgenos mejor caracterizados, conociéndose la estructura
primaria de la mayor parte de las variantes genéticas de as1-, as2-, b-
y k-caseína, b-lactoglobulina y a-lactoalbúmina(8-11).
En los últimos años han surgido estudios que ponen de relevancia la
asociación entre el reconocimiento de epítopos secuenciales y la
persistencia en la alergia a alimentos. Estos estudios, en el caso de la
leche han sido realizados en membranas SPOT®. Los trabajos que avalan
esta hipótesis son trabajos meramente descriptivos de poblaciones
homogéneas de pacientes con IgE específicas muy altas(12-18). No hay
ningún estudio publicado que explore la utilidad diagnóstica en una
población global de niños alérgicos a proteínas de leche de vaca en
seguimiento. Por lo tanto, el objetivo del presente estudio fue
determinar la especificidad y diversidad de anticuerpos IgE and IgG4
frente a epítopos secuenciales de αs1-, αs2-, β-, y k-caseinas, y b-lactoglobulina
mediante un microarray de péptidos.
MÉTODOS
Pacientes
Para llevar a cabo el objetivo del estudio se reclutaron pacientes en la
consulta de Alergia Infantil del Hospital Ramón y Cajal. Los pacientes
incluidos fueron todos previamente diagnosticados de alergia a proteínas
de leche de vaca mediada por IgE. El diagnóstico se realizó en base a
una anamnesis compatible con una reacción inmediata y la presencia de
pruebas cutáneas (PC) positivas con leche y fracciones (ALA, BLG y CAS)
y/o sIgE mediante CAP System FEIA [Phadia; Uppsala, Sweden]. Se
realizaron además provocaciones orales en el caso de historia clínica no
compatible. Siguiendo la práctica clínica habitual los pacientes fueron
puestos en dieta de exclusión durante 6 meses. Se re-evalúa a los
pacientes tras la dieta y se realizan de nuevo pruebas cutáneas e IgE
específica. Aquellos pacientes con una IgE frente a leche < 2.5 KU son
citados para una provocación oral con leche simple ciego, Gold Standard
para establecer si un paciente ha alcanzado la tolerancia.
En función del resultado de la prueba los pacientes se dividieron en dos
grupos: Tolerantes y Reactivos. El estudio fue aprobado por el CEIC
Hospital Ramón y Cajal.
Péptidos e inmunoensayo
Para este ensayo se sintetizaron comercialmente péptidos con una
longitud de 20 mer y 3 off-set (JPT Peptide Technologies) cubriendo la
secuencia primaria (lineal) de cada una de las proteínas estudiadas,
αs1-, α s2-, β- y k-caseínas, y β -lactoglobulina. De esta manera cada
péptido se diferencia del anterior en 3 aminoácidos hasta completar toda
la secuencia. Los péptidos se imprimen en triplicado en cristales epoxy
(ArrayIt) mediante un robot de impresión (NanoPrint™ Microarrayer 60,
TeleChem International, Inc.). Se incubaron los cristales con suero de
los pacientes (dilución 1:5) y como anticuerpo secundario se empleó un
cocktail 1:1 de anti-IgE (Phadia) y anti-IgG4 (Pharmigen) humano
monoclonal de ratón marcados con Alexa 546 y Alexa 647 (Molecular Probes)
respectivamente. Por último los cristales fueron escaneados (ScanArray®Gx,
PerkinElmer) y las imágenes almacenadas como archivos TIFF.
Análisis
La señal de fluorescencia de dos canales (para IgE e IgG4) fue
digitalizada (para cada spot fluorescente se obtiene un valor de
intensidad), normalizada, depurada y transformada logarítmicamente para
los análisis estadísticos posteriores.
Para determinar asociaciones estadísticas se utilizó el test exacto de
Fisher. Se consideró significativa una p<0,05.
Los péptidos con una intensidad (medida como Signal-Noise-Ratio, SNR) de
IgE/IgG4 por encima de 1 fueron considerados ‘péptidos
positivos’.Aquellos péptidos positivos que reconocían más del 75% de los
pacientes se consideraron epítopos. Se definieron los péptidos que
estadísticamente estaban asociados al grupo de pacientes con provocación
oral positiva.
Resultados
Finalmente se incluyeron en el estudio 31 pacientes entre enero de 2005
y febrero de 2006. La mediana de edad fue de dos años y cuatro meses
para ambos grupos (entre 1 y 6 años). El síntoma más frecuente en el
momento del diagnóstico fue la urticaria aislada seguida de urticaria y
síntomas digestivos. Se realizó provocación oral en 29 de los 31
pacientes. Los dos pacientes restantes tuvieron transgresiones
sintomáticas inmediatas con leche en su domicilio, el primero presentó
urticaria y el segundo urticaria y vómitos. Uno de los pacientes tuvo
una transgresión asintomática y la tolerancia fue posteriormente
confirmada en su domicilio. 16 pacientes tuvieron una provocación
positiva y 15 toleraron la leche. La IgE total mediana fue70 kU/l
(2->2000) en el grupo tolerante y 39 kU/l (17-323) en el grupo reactivo.
En cuanto a la IgE específica de leche fue 2.1 kUA/l (0.39-7.32) en los
tolerantes y 1.9 kUA/l (0.38-13) en los reactivos. No se encontraron
diferencias significativas en la presentación clínica, edad, sexo o
niveles de IgE entre ambos grupos (tabla 1).
Tabla
1. Características clínicas de ambos grupos (tolerantes y
reactivos).
|
|
TOLERANTES |
REACTIVOS |
|
Edad (mediana)* |
2 años 2 meses |
2 años 4 meses |
|
Sexo |
6 ♂ |
9 ♂ |
|
IgE total* |
70 kU/l (2->2000) |
39 kU/l (17-323) |
|
IgE leche*
|
2.1 kUA/l
(0.39-7.32) |
1.9 kUA/l
(0.38-13) |
|
Sintomas con la leche
|
urticaria |
8 (53.3%) |
8 (50%) |
|
urticaria + s.digestivos
|
5 (33.3%) |
3 (18.8%) |
|
urticaria + s.cutáneos
|
2 (13.3%) |
4 (25%) |
|
anafilaxia |
0 |
1 (6.3%) |
*Edad y valores
de IgE son en el momento de la provocación.
Tal y como se muestra
en la tabla 2 hay varias regiones en cada una de las proteínas
estudiadas, que fueron reconocidas por más del 75% de los pacientes
reactivos y fueron considerados por lo tanto epítopos. Posteriormente se
buscaron aquellos epítopos estadísticamente asociados con el grupo de
pacientes que tuvieron una provocación oral positiva con leche (tabla
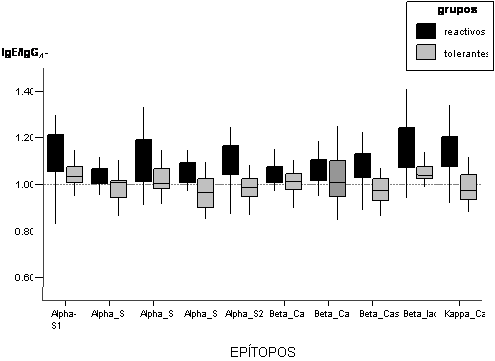
3). Se han encontrado 10 epítopos, a
s1 caseína: AA 28-50, a
s2 caseína: AA 1-20, 13-32, 67-86 y 181-207. ß-caseína: AA
25-50, 52-74 y 154-173. ß-lactoglobulina: AA 58-77.
k-caseína:
AA 34-53. La figura 1 representa, en un diagrama de cajas y bigotes,
estos diez epítopos (eje X) frente al ratio IgE/IgG4 SNR (eje Y). Se
muestran la mediana y el rango intercuartílico.
Tabla.2 Péptidos reconocidos por
más del 75% de los pacientes.
|
PROTEÍNA |
as1-
caseína |
as2-
caseína |
b
- caseína |
k
-caseína |
b
-lactoglobulina |
|
AA |
16-35 |
1-20 |
25-50 |
16-35 |
58-77 |
|
28-50 |
13-32 |
52-74 |
34-53 |
76-95 |
|
73-92 |
67-82 |
121-140 |
|
121-140 |
|
|
106-125 |
154-173 |
|
|
|
|
122-141 |
|
|
|
|
|
157-182 |
|
|
|
|
|
181-207 |
|
|
|
AA: aminoácido. En la tabla se muestra,
para cada proteína estudiada, la posición inicial y final del péptido en
la secuencia primaria (lineal) de la proteína.
Tabla 3. Epítopos asociados con el
grupo de pacientes con provocación positiva.
|
|
AA |
Reactivos
(n=16) |
Tolerantes
(n=15) |
p valor |
|
as1-
caseína
|
28-50 |
75% |
26.7% |
0.012 |
|
as2-
caseína |
1-20 |
75% |
13.3% |
0.001 |
|
|
13-32 |
75% |
26.7% |
0.012 |
|
|
67-86 |
75% |
33.3% |
0.032 |
|
|
181-207 |
75% |
20% |
0.004 |
|
b
- caseína |
25-50 |
75% |
33.3% |
0.032 |
|
|
52-74 |
81.3% |
26.7% |
0.004 |
|
|
154-173 |
75% |
33.3% |
0.032 |
|
k
-caseína |
34-53 |
87.5% |
40% |
0.009 |
|
b
-lactoglobulina
|
58-77 |
81.3% |
40% |
0.029 |
AA: aminoácido. Se muestra, para cada
epítopo definido, el porcentaje de reconocimiento entre el grupo de los
reactivos y para el grupo de tolerantes.
Figura 1.
Intensidad de los epítopos asociados con el grupo de pacientes reactivos
comparados con el grupo tolerante.
CONCLUSIONES
• Se trata del primer
trabajo de la literatura que ha explorado la utilidad diagnóstica de la
identificación de epítopos lineales mediante microarray en niños
alérgicos a proteínas de leche de vaca.
• Varias regiones han
sido identificadas como epítopos, mostrando que hay un patrón de
reconocimiento diferente entre los pacientes tolerantes y reactivos.
• Este estudio
representa un paso preliminar en el objetivo de encontrar mejores
instrumentos de diagnóstico para determinar si un paciente ha alcanzado
la tolerancia clínica a la leche de vaca.
References
(1) Lidholm J,
Ballmer-Weber BK, Mari A, Vieths S. Component-resolved diagnostics
in food allergy. Curr Opin Allergy Clin Immunol 2006; 6(3):234-40.
(2) Stein LD. Human
genome: end of the beginning. Nature 2004; 431(7011):915-6.
(3) Asero R,
Ballmer-Weber BK, Beyer K, Conti A, Dubakiene R, Fernandez-Rivas M
et al. IgE-mediated food allergy diagnosis: Current status and new
perspectives. Mol Nutr Food Res 2007; 51(1):135-47.
(4) Host A, Halken
S. A prospective study of cow milk allergy in Danish infants during
the first 3 years of life. Clinical course in relation to clinical
and immunological type of hypersensitivity reaction. Allergy 1990;
45(8):587-96.
(5) Host A,
Jacobsen HP, Halken S, Holmenlund D. The natural history of cow's
milk protein allergy/intolerance. Eur J Clin Nutr 1995; 49 Suppl
1:S13-S18.
(6) Sampson HA.
Food allergy. Part 2: diagnosis and management. J Allergy Clin
Immunol 1999; 103(6):981-9.
(7) Sampson HA.
Food allergy. Part 1: immunopathogenesis and clinical disorders. J
Allergy Clin Immunol 1999; 103(5 Pt 1):717-28.
(8) Farrell HM, Jr.,
Jimenez-Flores R, Bleck GT, Brown EM, Butler JE, Creamer LK et al.
Nomenclature of the proteins of cows' milk--sixth revision. J Dairy
Sci 2004; 87(6):1641-74.
(9) Wal JM, Pascal
G. Benefits and limits of different approaches for assessing the
allergenic potential of novel foods. Allergy 1998; 53(46 Suppl):98-101.
(10) Wal JM.
Structure and function of milk allergens. Allergy 2001; 56 Suppl
67:35-8.
(11) Wal JM. Cow's
milk proteins/allergens. Ann Allergy Asthma Immunol 2002; 89(6 Suppl
1):3-10.
(12) Ball G,
Shelton MJ, Walsh BJ, Hill DJ, Hosking CS, Howden ME. A major
continuous allergenic epitope of bovine beta-lactoglobulin
recognized by human IgE binding. Clin Exp Allergy 1994;
24(8):758-64.
(13) Busse PJ,
Jarvinen KM, Vila L, Beyer K, Sampson HA. Identification of
sequential IgE-binding epitopes on bovine alpha(s2)-casein in cow's
milk allergic patients. Int Arch Allergy Immunol 2002; 129(1):93-6.
(14) Chatchatee P,
Jarvinen KM, Bardina L, Vila L, Beyer K, Sampson HA. Identification
of IgE and IgG binding epitopes on beta- and kappa-casein in cow's
milk allergic patients. Clin Exp Allergy 2001; 31(8):1256-62.
(15) Chatchatee P,
Jarvinen KM, Bardina L, Beyer K, Sampson HA. Identification of IgE-
and IgG-binding epitopes on alpha(s1)-casein: differences in
patients with persistent and transient cow's milk allergy. J Allergy
Clin Immunol 2001; 107(2):379-83.
(16) Elsayed S,
Hill DJ, Do TV. Evaluation of the allergenicity and antigenicity of
bovine-milk alphas1-casein using extensively purified synthetic
peptides. Scand J Immunol 2004; 60(5):486-93.
(17) Jarvinen KM,
Chatchatee P, Bardina L, Beyer K, Sampson HA. IgE and IgG binding
epitopes on alpha-lactalbumin and beta-lactoglobulin in cow's milk
allergy. Int Arch Allergy Immunol 2001; 126(2):111-8.
(18) Vila L, Beyer
K, Jarvinen KM, Chatchatee P, Bardina L, Sampson HA. Role of
conformational and linear epitopes in the achievement of tolerance
in cow's milk allergy. Clin Exp Allergy 2001; 31(10):1599-606.
|
